最近AlphaFold开源,比较火,项目组也尝试进行复现,有些经验给大家分享一下,包括推理复现、训练复现、分布式训练复现等,今天先介绍一下推理的复现。
一、蛋白质结构预测背景与问题描述
蛋白质几乎参与所有生命现象,从催化化学反应的酶、到对抗病毒的抗体、以及作为信号物质的胰岛素。决定蛋白质功能的是由氨基酸序列折叠形成三维结构,各种蛋白质相互结合从而去影响生命现象。因此有“序列决定结构,结构决定功能。”的说法。
1972年诺贝尔化学奖的获奖演说中,克里斯蒂安·安芬森(Christian Anfinsen)提出了一个著名的假设:理论上,蛋白质的氨基酸序列应该完全决定它的结构。这一假设引发了一个长达50年的探索,即能够仅根据蛋白质的一级氨基酸序列来计算预测蛋白质的三维结构。然而,一个主要的挑战是,理论上一种蛋白质在形成最终的三维结构之前可以折叠的方式是天文数字。
基于高通量的测序技术的发展,使用DNA测序,从而测定蛋白质序列相对比较快速和便宜;然而,通过实验解析蛋白质结构则耗时长,成本高,难度大。常用的方法有X线晶体衍射图谱法,核磁共振法,冷冻电镜法等。
在UniProt数据库中,有超过2亿条以上的序列数据;然而在PDB数据中,仅有超过17万条以上(有部分重复)的结构数据。大部分的蛋白序列都没有结构数据。很自然的,如果能够通过序列预测结构,是很有科学价值的事情。
1994年,John Moult教授和Krzysztof Fidelis教授创立了CASP比赛。在CASP13以前,主要是以各种传统非端到端的预测的方法为主的,学术界也开始尝试引入深度学习,但离实验测得的结构差距较远。DeepMind在CASP13提出的Alpha Fold 1取得了显著的进步,CASP14参赛的全新实现的Alpha Fold 2更是在很多蛋白质的预测上接近实验数据。
为了后面的分享,我们再补充一些最必要的知识:
蛋白质的主链Backbone和侧链Sidechain构成。蛋白质的主链指的是那条肽链;侧链是每个氨基酸上决定不同氨基酸的部分(R)(氨基酸四个取代基NH2 ,COOH, H + 侧链R)。
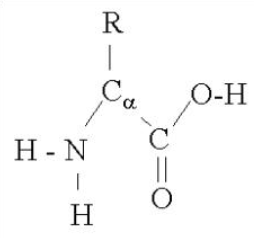
(说明:所有图片,来自于网络, 下同。)
氨基酸有20种,不同的氨基酸构成的序列,则为一级结构;部分子序列形成了相对固定的二级结构,如alpha-helix螺旋, beta-sheet带子;最终折叠成三级结构以及四级结构
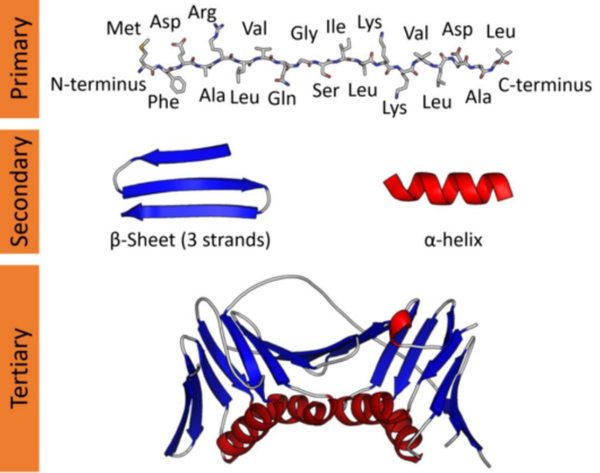
(注:图中的结构,为PyMOL等可视化软件卡通化的结果。)
Alpha Fold,CASP比赛,还有解蛋白质结构的生物学家,最终要解决就是通过蛋白质的氨基酸序列解析其3D结构。结构决定功能,最终在疾病治疗,生物制药等方面发挥作用。
为了理解算法,这些概念也比较重要:
Residue,残基,在蛋白质的序列中,氨基酸之间的氨基和羧基脱水成键,氨基酸由于其部分基团参与了肽键的形成,剩余的结构部分则称氨基酸残基。
Co-Evolution,共同进化,意思就是两个相关蛋白在进化过程中,一者发生突变,另一个蛋白对应的位点通常也会发生突变以继续保证二者之间的关联。做预测结构这个挺常用的,用来定位距离。
主链-侧链和各种角:
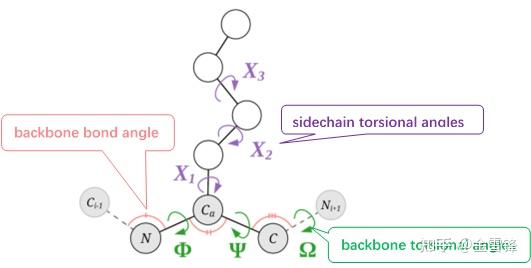
简单的说,这个问题就是:给定蛋白质的序列数据,预测出其3D结构。
其3D结构的表示,可以预测原子间距离,也可预测距离和角度,还可以直接预测原子的坐标等不同的做法;也可以先预测出中间结果残基的接触关系然后再预测出最终结果。
如果仅仅通过17万有结构的数据来直接训练,效果比较差。一般都会基于输入的序列,去寻找共进化的序列或序列的Embedding为原始输入的补充,这个过程即MSA多序列对齐;另外还会去搜索输入序列的同源的蛋白,这个过程即Template模板搜索。 除了Alpha Fold团队,比较知名的研究团队还有华盛顿大学的David Baker教授,密歇根大学张阳教授,芝加哥丰田计算技术研究所许锦波教授等。这次Science发表的RoseTTAFold算法的David团队,在算法中还引入了二级结构的搜索结果。
(说明:关于蛋白质相关生物问题描述,如有错误,在所难免。)
二、AlphaFold2算法结构
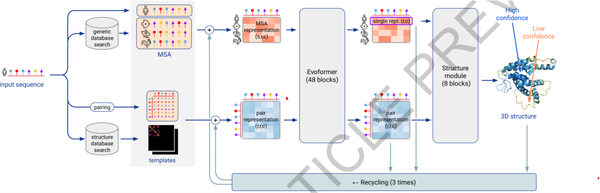
Alpha Fold的开源,在论文、补充材料和代码中,给出了非常详细的信息,除了没有训练用的代码和训练数据(如MSA和Template数据,需要用相关工具从原始数据上生成)。
上图输入侧的四条线,分别表示:原始的输入序列;通过hhblits/JackHMMER搜索的MSA(序列-残基);搜索的结构和序列产生的Pair(残基-残基);通过HHsearch搜索的Template。
在算法部分,为主干为EvoFormer,结构部分为等变Transformer,且在两部分做“迭代”提升。DeepMind认为,EvoFormer核心是MSA 内交换信息的新机制,并且其配对表示允许直接推理空间和进化关系,而Equivariant Attention主要是能适应每个残基的旋转和平移,更精细的优化主链侧链里的结构。
在CASP14后基于DeepMind透露的有限的信息,大家在尝试复现AlphaFold2的各种技术点,对比这些技术点:
- 算法中广泛使Attention,各种Attention变体如Axial Attention,引入等变Transformer,Pairwise Representation,Graph表达(残基作为节点,残基间关系作为边),端到端直接预测坐标或等价的表示,充分利用MSA和Template的共进化、同源等信息等;
- RoseTTAFold也包含逐步优化的思想(在Baker的RoseTTAFold算法中,近似的迭代优化为Iterative Feature Extractor,其迭代块为Transformer,ResNet,SE3。)
- 开源的AlphaFold2的联合从Label和Unlabeled数据中学习这部分,是复现工作没有涉及的。 其做法为:先用17万中有X序列-Y坐标的先训练,然后用初训练的网络对Uniclust30更多的(350,000)只有X序列的做预测和评估,对预测的好的(high-confidence subset)也作为训练样本,然后做数据增强(cropping and MSA subsampling),最终取得更好的效果,该技术点为noisy student self-distillation,在很多其他场合被使用。
三、AlphaFold2 / RoseTTAFold算法复现
AlphaFold的训练部分的复现,需要几部分的必要工作:
1)大数据集的下载(数百G)和MSA/Template的生成,生成非常耗时,这个需要大量CPU服务器并行,生成的数据在数T以上;以Sequence长度PDB数据库中50-60的短序列的MSA生成单服务器,hhblits缺省参数,都在1周左右。
2)基于jax,haiku的训练代码的实现和并行训练。
3)初训练和扩展数据集的形成。
后面两块AlphaFold2还没有看到看到开源实现。
如果您对从零完整复现训练感兴趣,可以关注我们后续复现工作的分享。
这里,我们先分享,不基于docker的,复现AlphaFold开源的推理,RoseTTAFold的推理。
1、Alpha Fold 2的host版本推理,Ubuntu18.04+CUDA10.1
- 下载数据
apt-get update && apt-get -y install aria2
nohup ~/alphafold/scripts/download_pdb_mmcif.sh /root/alphafold/data/pdb_mmcif/ & #这部分需要rsync和aria,是很多分散的文件
wget other datasets- 构建host environment, not docker
cd ~/
git clone https://github.com/deepmind/alphafold.git
cd alphafold
pip install dm-haiku #jax之上最流行的NN库
pip install simtk
pip install openmm
pip install absl-py==0.13.0
#下面部分为DeepMind的docker版本中在docker内安装的软件
curl -fsSL https://mirrors.aliyun.com/nvidia-cuda/ubuntu1804/x86_64/7fa2af80.pub | apt-key add -
echo "deb https://mirrors.aliyun.com/nvidia-cuda/ubuntu1804/x86_64/ /" > /etc/apt/sources.list.d/cuda.list
apt update
DEBIAN_FRONTEND=noninteractive apt-get install -y build-essential cmake git hmmer kalign tzdata wget cuda-command-line-tools-10-1
rm -rf /var/lib/apt/lists/*
git clone --branch v3.3.0 http://github.com/soedinglab/hh-suite.git /tmp/hh-suite
mkdir /tmp/hh-suite/build && cd /tmp/hh-suite/build
cmake -DCMAKE_INSTALL_PREFIX=/opt/hhsuite ..
make -j 4 && make install
ln -s /opt/hhsuite/bin/* /usr/bin
#rm -rf /tmp/hh-suite
wget -q -P /tmp https://repo.anaconda.com/miniconda/Miniconda3-latest-Linux-x86_64.sh
bash /tmp/Miniconda3-latest-Linux-x86_64.sh -b -p /opt/conda
#rm /tmp/Miniconda3-latest-Linux-x86_64.sh
#注意一定要使用Python3.8,后面openmm的patch也是针对该版本,这是个坑
#DeepMind的在Ubuntu20+CUDA11.0,使用Ubuntu18.04+CUDA10.1也可以,需要注意修改脚本中所有10.1、101,10-1的地方。
export PATH="/opt/conda/bin:$PATH"
conda update -qy conda
conda install -y -c conda-forge openmm=7.5.1 cudatoolkit==10.1.243 pdbfixer pip- 因为是推理,不修改网络结构,直接使用DeepMind公开的模型
wget -q -P ~/alphafold/alphafold/common/ https://git.scicore.unibas.ch/schwede/openstructure/-/raw/7102c63615b64735c4941278d92b554ec94415f8/modules/mol/alg/src/stereo_chemical_props.txt
pip3 install --upgrade pip
pip3 install -r ~/alphafold/requirements.txt
pip3 install --upgrade jax jaxlib==0.1.69+cuda101 -f https://storage.googleapis.com/jax-releases/jax_releases.html
cd /opt/conda/lib/python3.8/site-packages
patch -p0 < ~/alphafold/docker/openmm.patch
cd ~/alphafold
wget https://storage.googleapis.com/alphafold/alphafold_params_2021-07-14.tar && mkdir && tar params -xvf alphafold_params_2021-07-14.tar -C params- 从casp14下载T1050蛋白的序列
wget -o /dev/null -O T1050.fasta 'https://www.predictioncenter.org/casp14/target.cgi?target=T1050&view=sequence'- 预测也需要搜索MSA和Template,需要对应指定的数据文件路径
export DATA_DIR='/root/alphafold/data/' && export OUTPUT_DIR='/root/alphafold/output/'- 开始预测
python run_alphafold.py --fasta_paths=${DATA_DIR}/fasta_path_0/T1050.fasta --uniref90_database_path=${DATA_DIR}/uniref90_database_path/uniref90.fasta --mgnify_database_path=${DATA_DIR}/mgnify_database_path/mgy_clusters.fa --uniclust30_database_path=${DATA_DIR}/uniclust30_database_path/uniclust30_2018_08 --bfd_database_path=${DATA_DIR}/bfd_database_path/bfd_metaclust_clu_complete_id30_c90_final_seq.sorted_opt --pdb70_database_path=${DATA_DIR}/pdb70_database_path/pdb70 --data_dir=${DATA_DIR}/data_dir/data --template_mmcif_dir=${DATA_DIR}/template_mmcif_dir/mmcif_files --obsolete_pdbs_path=${DATA_DIR}/obsolete_pdbs_path/obsolete.dat --output_dir=${OUTPUT_DIR}/output --model_names=model_1,model_2,model_3,model_4,model_5 --max_template_date=2020-05-14 --preset=full_dbs --benchmark=False --logtostderr2、RoseTTAFold的推理,Ubuntu18.04+CUDA10.1
- 安装相关的Python包
###pip
#dgl: https://www.dgl.ai/pages/start.html
cat /usr/local/cuda/version.txt
python --version
pip install dgl-cu101
pip install torch_geometric torch_sparse torch_scatter- 安装序列对齐和模板搜索软件
#hh-suite
wget https://github.com/soedinglab/hh-suite/releases/download/v3.2.0/hhsuite-3.2.0-AVX2-Linux.tar.gz --no-check-certificate
tar xvfz hhsuite-3.2.0-AVX2-Linux.tar.gz
mkdir hhsuite
mv bin hhsuite
mv data hhsuite
mv scripts hhsuite
mv LICENSE hhsuite
mv README hhsuite
# Backer版本,使用hhsuite3.2.0不会crash
#nibi-blash (legacy)
wget ftp://ftp.ncbi.nih.gov/blast/executables/legacy.NOTSUPPORTED/2.2.26/blast-2.2.26-x64-linux.tar.gz
tar -xzvf blast-2.2.26-x64-linux.tar.gz
#gcc5.4/g++5.4
apt install gcc-5 g++-5
update-alternatives --install /usr/bin/gcc gcc /usr/bin/gcc-5 40
update-alternatives --install /usr/bin/g++ g++ /usr/bin/g++-5 40
#git clone https://github.com/sparsehash/sparsehash
apt-get install libsparsehash-dev
### official: http://wwwuser.gwdg.de/~compbiol/data/csblast/releases/csblast-2.2.3_${platform}.tar.gz
git clone https://github.com/soedinglab/csblast
cd csblast/src
make csblast
make csbuild
make csclust
make cssgd
make cstrainset
make cstranslate- 使用官方提供的权重
wget https://files.ipd.uw.edu/pub/RoseTTAFold/weights.tar.gz
tar xfz weights.tar.gz- 下载RoseTTA的软件
git clone https://github.com/RosettaCommons/RoseTTAFold
cd RoseTTAFold- 修改多个脚本
###modify script,主要是环境配置的修改
修改run_e2e_ver.sh脚本
#set -e
#mkdir -p $WDIR/log
#conda activate RoseTTAFold
#Backer版本,有端到端版本,还有多步的版本;对步的版本,会先预测出多组候选距离与朝向,然后做精化,最终评估选择出最好的结果
修改run_pyrosetta_ver.sh脚本
#set -e
#mkdir -p $WDIR/log
#conda activate RoseTTAFold
#conda deactivate
#conda activate folding
修改input_prep/make_msa.sh
#-d $MYDB #just use one db for test
修改input_prep/make_ss.sh中的修改相关路径,参考如下:
DATADIR="/root/data/psipred/soft/psipred/data"
/root/data/psipred/soft/blast-2.2.26/bin/makemat -P $ID- 准备数据
cd <workspace>
##/root/RoseTTAFold/bfd/
ln -s /root/data/hh/data/UniRef30_2020_06/ .
ln -s /root/data/baker/pdb100_2021Mar03/ .
ln -s /root/data/psipred/soft/csblast/ .
mv ./csblast ./csblast-2.2.3
ln -s /root/data/baker/weights/ .- 执行shell脚本,开始预测,shell脚本任选一个或者都执行
#端到端预测,效果略差
run_e2e_ver.sh
#pipeline多步预测
run_pyrosetta_ver.sh如上,最直白最简单的介绍了蛋白质结构预测问题和相关比赛,对nature和science最新发表的两篇相关前沿论文,做了简单的技术点分析,也分享了我们在复现过程中的一些经验。 如果您对完整的复现包含训练感兴趣,可以关注我们后续的分享,包含训练过程,分布式版本,进一步调优等工作。
原文:知乎
作者:金雪锋
推荐阅读
更多嵌入式AI技术干货请关注嵌入式AI专栏。


